Bacteriófago

Como bacteriófagos o simplemente fagos ( fago singular , el ; del griego antiguo βακτήριον baktērion , Chopsticks 'y φαγεῖν phagein , eat') se refiere a varios grupos de virus que se especializan en bacterias como células huésped . Según la especificidad del hospedador, los fagos se dividen en grupos taxonómicos , por ejemplo bacteriófagos coli , estafilococos , difteria o Salmonella . Con un número estimado de 10 30 viriones en toda el agua de mar, los fagos son más comunes que cualquier tipo de ser vivo y forman lo que se conoce como virioplancton , incluidos muchos cianófagos (bacteriófagos de cianobacterias ).
Tradicionalmente, los virus de las arqueas ( arqueovirus, virus de arqueas, arqueovirus ) a veces todavía se denominan fagos (bacterianos), lo que todavía recuerda a principios de la década de 1970, cuando las arqueas aún no se diferenciaban de las bacterias y fagos con estructura de cabeza. investigados ( Caudoviricetes ), que infectan en parte bacterias y en parte arqueas.
Tenga en cuenta: los virus no son seres vivos porque no tienen su propio metabolismo . Algunos científicos los llaman "cercanos a la vida".
historia
El efecto de los fagos fue descrito por primera vez en 1917 por el franco-canadiense Félix Hubert d'Hérelle . Aunque el inglés Frederick Twort ya había observado procesos de descomposición en cultivos de estafilococos dos años antes , que se remontan a la acción de los bacteriófagos, su publicación fue prácticamente ignorada. D'Hérelle es, por tanto, uno de los descubridores de los bacteriófagos, los llamados "comedores de bacterias", junto a Frederick Twort. Sin embargo, deben su nombre y su descubrimiento a d'Hérelle. Paralelamente a d'Hérelle, el microbiólogo alemán Philalethes Kuhn postuló la existencia de parásitos bacterianos basándose en observaciones de cambios en cultivos bacterianos en determinadas condiciones. Se refirió a esto como Pettenkoferien y vio el “microbio invisible que contrarresta la disentería” descrito por d'Hérelle como un caso especial de estos parásitos. Como resultó más tarde, sus observaciones no se basaron en la existencia de un parásito bacteriano, sino solo en cambios en la forma de la bacteria que estaba estudiando.
D'Hérelle imaginó al bacteriófago como un " ser vivo corpuscular ultravisible " que existe en una forma básica y que se adapta a diferentes huéspedes, es decir, bacterias. De hecho, hasta donde sabemos hoy, los bacteriófagos son virus altamente especializados que están unidos a un huésped específico. Incluso si hablamos de huéspedes en este contexto, según la definición actual, los bacteriófagos, ya que como virus, no son seres vivos, no son parásitos . El primer fago que se examinó fueron siete fagos de la bacteria Escherichia coli . Fueron nombrados por Max Delbrück en el orden de su descubrimiento como Tipo ( Tipo inglés ) 1 (T1), Tipo 2 ( T2 ) y así sucesivamente. La clasificación taxonómica actual de estas cepas de fagos según ICTV con especies y familias es la siguiente:
- T1: Especie Escherichia virus T1 , Drexlerviridae
- T2: Especie Escherichia virus T4 (alias T-incluso fagos ), Myoviridae
- T3: Especie Escherichia virus T3 , Autographiviridae
- T4: Tipo de cepa de la especie Escherichia virus T4 .
- T5: Especie Escherichia virus T5 , Demerecviridae
- T6: también especie Escherichia virus T4
- T7: Especie Escherichia virus T7 , Autographiviridae
Estos siete fagos a veces se agrupan bajo el nombre colectivo de fagos T (en. Fagos T ), pero esto no representa un grupo familiar ( taxón ). En cambio, estos bacteriófagos serán asignados a las familias indicadas anteriormente por el ICTV (a partir de enero de 2021) después de algunos turnos. Solo los fagos con un número de tipo par ( fagos T-pares ) resultaron estar estrechamente relacionados por casualidad, por lo que una especie se estableció como taxón para la ICTV. Los tipos con números impares ( fagos T-impares / T-desiguales ) no forman un taxón. Sin embargo, todos estos tipos de fagos son una estructura de cabeza a cola juntos, lo que ven como representantes de las características del orden de virus Caudovirales . Posteriormente, esta práctica fue parcialmente continuada por otros autores al nombrar otros caudovirus (por ejemplo, " T12 ", sugerencia, sin asignación familiar).
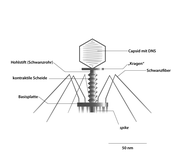
construcción

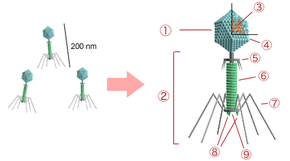
2 cola
3 ácido nucleico
4 cápside
5 collar
6 vaina
7 fibra de la cola
8 puntas
9 placa base
La forma de los bacteriófagos con una estructura de cabeza a cola (orden Caudovirales ) se dilucidó principalmente en los fagos de la serie T (serie T) de Escherichia coli . El bacteriófago T2 consta de una cabeza poliédrica de 100 nm de longitud con una cola de aproximadamente la misma longitud. Los bacteriófagos se agrupan taxonómicamente según su morfología , genoma y hospedador . Se hace una distinción entre fagos de ADN con ADN monocatenario, los denominados fagos ssDNA (del inglés monocatenario ), y aquellos con ADN bicatenario, los denominados fagos dsDNA (del inglés bicatenario ). Los fagos de Escherichia coli de la serie T tratados aquí a modo de ejemplo pertenecen al último grupo.
Los fagos T ya mencionados anteriormente (como el género Tequattrovirus con la especie Escherichia virus T4 ), junto con otros miembros del orden Caudovirales, se distinguen de otros bacteriófagos por una estructura relativamente compleja con una “estructura cabeza-cola”: Básicamente constan de una placa base (9), un dispositivo de inyección (dispositivo de inyección o cola, 2) y una cabeza (1), formada por la denominada cápside (4) y el ácido nucleico que contiene ( genoma , 3). Los módulos de cabeza e inyector / cola están conectados por un cuello (collar, 5). La placa base (que, al igual que la cápside y el dispositivo de inyección, está formada por proteínas ) está cubierta con fibras de la cola (7) y picos (8), que se utilizan para la adsorción en la pared de la célula huésped . El aparato de inyección consta de un tubo delgado (tubo de cola, 6) a través del cual se inyecta el ácido nucleico del fago (3) en la célula huésped. El tubo está envuelto en una vaina de cola contráctil que se contrae durante la inyección. La cápside está compuesta por 152 capsómeros con simetría icosaédrica y contiene el ADN del fago. Debido a esta estructura, los fagos del género Tquattrovirus (familia Myoviridae ) se encuentran entre los virus estructuralmente más complejos.
Los fagos con ADN monocatenario, por otro lado, suelen ser pequeños, esféricos y sin cola o filamentosos. Los fagos de ARN que también aparecen suelen consistir (si se describen hasta este momento) en una envoltura de proteína que encierra una molécula de ARN monocatenaria. El diámetro de estos fagos ronda los 25 nm, lo que los convierte en uno de los fagos más pequeños.
- Diferentes fagos
Fago T2 ; Cápside de corte axial
Lysehof de Bacillus phage gamma en Bacillus anthracis , a la derecha colonia única no infectada
Synechococcus fago S-PM2 del agua de mar
Fago lambda ( virus Escherichia lambda ); una sección transversal esquemática
Multiplicación
En ausencia de un metabolismo propio, los virus requieren un huésped para la reproducción , en el caso de los bacteriófagos, una célula bacteriana viva adecuada. La reproducción se puede dividir en cinco fases:
- Adsorción a receptores específicos de la pared celular : durante la adsorción, los extremos de las fibras de la cola se acoplan a moléculas adecuadas ( receptores ) en la superficie de la bacteria.
- Inyección del ácido nucleico del fago en la célula huésped: el propio ácido nucleico del fago , ADN o ARN , entra en la bacteria. Las proteínas ahora sin función de la envoltura del fago vacía permanecen afuera en la superficie de la bacteria.
- Fase de latencia: durante esta fase, no se pueden detectar fagos en la bacteria. La transcripción de la genoma viral , la traducción de la viral ARNm y la replicación del ácido nucleico viral ahora comenzar . Este proceso tarda como máximo unas horas.
- Fase de producción: una vez que los genes del fago se han activado en una secuencia fija, se forman todos los componentes del virus, las proteínas de la envoltura y las fibras de la cola.
- Fase de madurez: en esta fase de morfogénesis , tiene lugar el ensamblaje para formar partículas de fagos maduros. Primero, se forma una parte de la cabeza, la cápside . Las proteínas del interior sirven como marcadores de posición y luego son reemplazadas por el ácido nucleico del fago que penetra en la cápside. Los hilos de ácido nucleico adoptan una forma que ahorra espacio, como una bola de lana.
- Liberación: las partículas de virus terminadas se liberan por disolución enzimática de la célula huésped. La lisozima , que fue formada por la bacteria reprogramada, disuelve la pared celular bacteriana murina . La célula estalla y se liberan unos 200 fagos infecciosos.
En el caso de algunos tipos de fagos, la reproducción no siempre procede de acuerdo con el esquema lítico descrito anteriormente . En el caso de los fagos templados , se hace una distinción entre ciclos de reproducción lisogénica y lítica o ciclos de infección. En un ciclo lisogénico, el ADN del fago se incorpora al cromosoma de la bacteria, creando un profago . Con cada división celular subsiguiente, los genes del fago y los de la bacteria se duplican y se transmiten juntos. Este ciclo puede conducir posteriormente al ciclo lítico.
Fagos gigantes

Los fagos de ADN bicatenario con un tamaño de genoma de más de 540 kbp se denominan megáfagos, mientras que los más pequeños con más de 200 kbp se denominan fagos jumbo. En 2018/2019, los autores examinaron las heces de personas en Bangladesh y Tanzania, así como babuinos en África y cerdos en Dinamarca. Las muestras contenían bacterias del género Prevotella ( Prevotellaceae ), que fueron infectadas por varios megafagos de dsDNA, que los autores denominaron “ fagos Lak ” (en honor a la ubicación de Laksam Upazila , Bangladesh). Los fagos encontrados se designaron (provisionalmente) como Lak-A1, Lak-A2, Lak-B1 a Lak-B9 y Lak-C1. Podría haber una relación filogenética laxa con "Sphingomonas Phage PAU" (este fago gigante infecta bacterias de la especie Sphingomonas paucimobilis , Sphingomonadaceae ) y, por tanto, con la familia de fagos Myoviridae . Los autores llegan a la conclusión de que los "fagos lac" son "miembros del microbioma intestinal generalizados, pero previamente pasados por alto".
En febrero de 2020, Basem Al-Shayeb y sus colegas publicaron un análisis que continúa estas investigaciones. En él trazan el límite de megafagos a 500 kpb (lo que obviamente significa pares de bases en el caso de doble hebra y bases o nucleótidos en el caso de hebra simple). Los autores prefieren considerar todos los fagos con más de 200 kpb (es decir, fagos gigantes y megáfagos) como " fagos enormes ingleses " (traducidos aquí como fagos gigantes). Los autores identificaron una serie de diez clados entre este grupo , para los que sugirieron los siguientes nombres: "Kabirphage", "Mahaphage" (incluido el grupo de los " fagos Lak "), "Biggiephage", "Dakhmphage", "Kyodaiphage" , "Kaempephage", "Jabbarphage", "Enormephage", "Judaphage" y "Whopperphage" (todos los nombres se refieren a "enorme" o " enorme " en los diferentes idiomas de los autores). A través de sus análisis metagenómicos de varias muestras, pudieron identificar 351 secuencias de fagos de dsDNA, de las cuales solo 3 tenían menos de 200 kpb. El genoma más grande tenía 735 kpb de longitud (un Mahaphage, que aparentemente es un nuevo récord; el anterior tenía 596 kpb); los fagos no gigantes ordinarios tienen un promedio de solo 52 kpb. Algunos fagos gigantes parecen utilizar un código genético no estándar en el que el codón de parada UAG codifica un aminoácido . Los huéspedes son (en su mayoría) bacterias de Firmicutes o Proteobacteria , pero también - como en el caso de los miembros del grupo Mahaphage con los "fagos Lak" - Bacteroidetes . El genoma codifica los ARNt además de las proteínas comúnmente utilizadas en los fagos . Los fagos también interactúan en el sistema CRISPR / Cas (ver CRISPR , método CRISPR / Cas , edición del genoma ): todos los tipos principales del sistema estaban representados, pero la mayoría de los fagos parecían usar las proteínas Cas del anfitrión para generarse a sí mismos para protegerse. Además, los fagos parecían ayudar al sistema inmunológico CRISPR de los anfitriones a luchar contra los fagos competidores. Algunos fagos que infectan a Pseudomonas también codifican anti-CRISPR (Acrs) y proteínas que forman un compartimento similar a un núcleo en el que el fago puede replicar su genoma de forma más independiente del huésped (ver Viroplasma ). Los autores ven su trabajo como una prueba más de la distribución mundial de los fagos gigantes. Encontraron evidencia de que los fagos migraron entre diferentes huéspedes y ecosistemas, lo que tiene implicaciones para la propagación de genes de resistencia a toxinas y antibióticos . Sus herramientas CRISPR podrían utilizarse en el futuro para mejorar las "tijeras genéticas" CRISPR / Cas y ampliar su funcionalidad.
Otro fago gigante es el fago Megasphaera A9 (alias Huge Phage A9 ), que no debe confundirse con el fago Brochothrix A9 (especie Brochothrix virus A9 , Herelleviridae ).
Fagos sin cola
Durante mucho tiempo, la investigación solo ha considerado miembros del orden Caudovirales , cuyos representantes son fagos (virus bacterianos y arqueales) con una estructura de cabeza a cola. Sólo recientemente los fagos "sin cola" se han convertido en objeto de investigación. Algunos representantes son:
- Solicite Tubulavirales (bacteriófagos filamentosos) y otros. con la familia Inoviridae
- Familia Finnlakeviridae (ssDNA, con la especie Flavobacterium virus FLiP alias Phage FLiP )
- Familia " Autolykiviridae " (dsDNA, propuesto)
- Especie Planktothrix-Phage PaV-LD ( Cyanophage PaV-LD )
crAssphages y Gubaphages
Camarillo-Guerrero, Almeida, et al. 2019/2020 describen los resultados de sus análisis metagenómicos de la flora intestinal humana con respecto a los bacteriófagos. Lo convierten en un nuevo clado, llamado "Gubaphagen" ( fago inglés G ut Ba cteroidales , clado Gubaphage ) (con dos géneros, Bacteroides infectados con G1 y Parabacteroides [s] infectados con G2 ), que según el fago craso con aproximadamente 10 Los géneros ( fagos similares a crAss , miembros propuestos originalmente del orden Caudovirales , familia Podoviridae , con aproximadamente 10 géneros) representan el segundo virus más común (es decir, bacteriófagos) en este entorno. Las características de los Gubaphages recuerdan a las de "p-crAssphage".
Mientras tanto, se ha propuesto dar a los crAssphages su propio orden Crassvirales dentro de la clase Caudoviricetes . Debido a su similitud con los crAssphages, los Gubaphages probablemente también serían miembros de los Crassvirales (o al menos los Caudoviricetes ).
Halovirus
Bajo el nombre informal (no taxonómico), los halovirus ( halovirus en inglés ) se clasifican como fagos que atacan a las bacterias halófilas y arqueas. Además del género Myohalovirus ( Caudovirales : Myoviridae ) con la especie Halobacterium virus phiH confirmada por el ICTV y la especie propuesta "Halorubrum phage HF2", estas son especies sin clasificar y aún sin confirmar "HF1", "HCTV-1", " 2 "y" 5 "," HGTV-1 "," HHTV-1 "y" 2 "," HRTV-4 "," 5 "," 7 "y" 8 "( caudovirales )," HSTV-1 "( caudovirales : Podoviridae ) y "2" ( Caudovirales : Myoviridae ), "HVTV-1" ( Caudovirales : Siphoviridae ), "Halovirus VNH-1" ("VNH-1", Fuselloviridae ) y "Haloferax tailed virus 1" (HFTV1, Caudovirales ).
Magrovirus
Las arqueas marinas de Euryarchaeota se clasifican como Marine Group ( English Marine Group ) II (MG-II, que consta de MG-IIa a MG-IId), III (MG-III) y IV (MG-IV) - the Marine El grupo I (MG-I) denota arqueas marinas de Thaumarchaeota .
La denominación no taxonómica Magroviruses ( en inglés magroviruses , virus marinos Grupo II ) se utiliza para fagos que parasitan Clasificar Euryarchaeota del primer grupo mencionado MG-II. Se trata de virus dsDNA con un tamaño de genoma de 65-100 kpb con una estructura de la cabeza a la cola: "Magrovirus A", "Magrovirus B1" y "B2", así como "Magrovirus C" y (sospechoso) "Magrovirus D". ".
Áreas de aplicación
Los fagos han encontrado una amplia gama de aplicaciones en medicina , biología , ciencias agrícolas , especialmente en el área de la ingeniería genética . Debido a su especificidad de hospedador, los fagos se utilizan en medicina para determinar patógenos bacterianos . Este procedimiento se llama lisotipo . Debido a la frecuencia cada vez mayor de resistencias múltiples a los antibióticos , actualmente se están llevando a cabo investigaciones intensivas sobre el uso de bacteriófagos como sustitutos de antibióticos en la medicina humana (ver: terapia con fagos ). Los problemas surgen aquí por la escasa estabilidad de los fagos en el cuerpo, ya que son eliminados como cuerpos extraños en muy poco tiempo por los fagocitos . Felix d'Hérelle (ver arriba) descubrió esta aplicación de fagos para el tratamiento de infecciones bacterianas mucho antes del descubrimiento de la penicilina y los antibióticos . Más tarde, sin embargo, con la introducción de la quimioterapia con antibióticos, la terapia con fagos se consideró poco práctica y se olvidó. En 1934, D'Hérelle fundó el Instituto Eliava de Investigación de Fagos junto con el microbiólogo georgiano Georgi Eliava en la República Socialista Soviética de Georgia , que todavía existe en la actualidad. Hoy en día, la terapia con fagos para infecciones bacterianas resistentes a la terapia se lleva a cabo allí y en el Instituto Ludwik Hirszfeld de Inmunología y Terapia Experimental en Wroclaw (parte de la Academia Polaca de Ciencias ) . En Alemania, el uso con fines terapéuticos aún no está permitido.
Las aplicaciones en la producción de alimentos son diversas; Por ejemplo, se utiliza un spray de fagos para envasar salchichas o rebanar queso.
En ingeniería genética , los fagos templados se utilizan como vectores (por ejemplo, el fago λ ). Para ello, los fagos se preparan de tal forma que los genes que provocan la virulencia se extraen de su genoma y se reemplazan por genes que son de interés para fines de ingeniería genética, como los genes necesarios para la producción de insulina . A continuación, estos fagos modificados se ponen en contacto con bacterias adecuadas, por ejemplo, E. coli . Después de comprobar si el gen deseado se ha integrado en el material genético del genoma bacteriano (se utilizan resistencias a antibióticos expresadas por genes que están conectadas a los genes deseados que se van a clonar ), las células bacterianas modificadas se pueden cultivar más y la insulina se puede producir en este caso se puede aislar. De manera similar, los fagos se utilizan en tecnología agrícola para transducir ciertos genes en cultivos . Una aplicación importante en bioquímica es la presentación de fagos para la identificación de socios de unión, p. B. en el aislamiento de nuevos principios activos.
Sin embargo, la transformación de ADN libre, que hoy en día se utiliza principalmente para la transferencia a células bacterianas, es más fácil que utilizar fagos .
Los fagos y componentes se utilizan para eliminar la contaminación microbiana en los alimentos (por ejemplo, mediante separación magnética por afinidad ), así como en muestras de laboratorio contaminadas con endotoxinas . Además, existen aplicaciones de diagnóstico humano, especialmente en el área clínica para la descolonización de gérmenes patógenos hospitalarios como el MRSA . Las proteínas del fago se pueden optimizar para la aplicación respectiva mediante el diseño de proteínas .
Posible daño económico
Los bacteriófagos pueden causar daños dondequiera que los procesos bacterianos sirvan a los humanos y sean deseables. La infección de bacterias del ácido láctico (BAL) por fagos de la leche cruda es la causa más común de actividad enzimática reducida o ausente en cultivos iniciadores para la producción de queso o leche cuajada .
clasificación
Los virus procarióticos (virus bacterianos y arqueales , "bacteriófagos") no forman un grupo familiar cerrado ( taxón ). Para muchos grupos de fagos todavía hay nombres informales después de sus anfitriones (ver más arriba), p. Ej. B. Cianófagos ( cianobacterias ), Colifagos (Colibacterium E. coli ), estos tampoco son grupos relacionados.Otra especialidad son los virus satélite cuyos virus auxiliares son los bacteriófagos, a veces se les llama fagos satélite. Un ejemplo es el “fago P4 de Escherichia”, que requiere el colifago P2 ( Myoviridae , género Peduovirus ) como virus auxiliar.
Clasificación de Baltimore
Según la clasificación de Baltimore , los fagos se pueden agrupar según la estructura de su genoma de la siguiente manera:
- Bacteriófagos de dsDNA:
- Familia: Ackermannviridae
- Familia: Autographiviridae
- Familia: Chaseviridae
- Familia: Demerecviridae
- Familia: Drexlerviridae
- Familia: Herelleviridae
- Familia: Myoviridae
- Familia: Podoviridae
- Familia: Siphoviridae
- Familia: Tectiviridae
- Familia: Corticoviridae
- Familia: Plasmaviridae
- Familia: Lipothrixviridae
- Familia: Rudiviridae
- Familia: Fuselloviridae
- Familia: Halspiviridae (con género: Salterprovirus y especie Salterprovirus His1 alias His 1 virus )
- Familia: Guttaviridae
- Familia: Bicaudaviridae
- Familia Thaspiviridae (con el género Nitmarvirus y la especie Nitmarvirus NSV1 alias Nitrosopumilus maritimus virus 1 )
- Familia "Autolykiviridae" (sugerido, ver arriba)
- Bacteriófagos de ssDNA:
- Familia Finnlakeviridae
- Familia: Inoviridae - división propuesta basada en datos del metagenoma: "Amplinoviridae", "Protoinoviridae", "Photinoviridae", "Vespertilinoviridae", "Densinoviridae" y "Paulinoviridae"
- Familia: Plectroviridae
- Familia: Microviridae
- Caso especial
- Familia Pleolipoviridae (con el género Gammapleolipovirus y la especie Gammapleolipovirus His2 alias His 2 virus , Haloarcula virus His2 )
- Bacteriófago dsRNA:
- Familia: Cystoviridae
- Bacteriófagos de ssRNA:
- Familia: Fiersviridae (también conocido como Leviviridae )
Clasificación taxonómica según ICTV
En el sistema de taxonomía de virus según el Comité Internacional de Taxonomía de Virus (ICTV) , los fagos se pueden encontrar en los siguientes grupos taxonómicos:
| zona | pedido | familia | morfología | Genoma | Ejemplos de |
|---|---|---|---|---|---|
| Riboviria | Levivirales | Leviviridae | un envuelto , isométrico | ssRNA , lineal | MS2 , Qβ |
| Mindivirales | Cystoviridae | envuelto, esférico | dsRNA , segmentado | Phi6 | |
| Varidnaviria | Belfryvirales | Turriviridae | envuelto, isométrico | dsDNA , lineal | STIV1 |
| Halopanivirales | Sphaerolipoviridae | envuelto, isométrico | dsDNA, lineal | Fago SH1 | |
| Kalamavirales | Tectiviridae | descubierto, isométrico | dsDNA, lineal | PRD1 | |
| Vinavirales | Corticoviridae | descubierto, isométrico | dsDNA, circular | PM2 | |
| Duplodnaviria | Caudovirales | Ackermannviridae | cola descubierta, contráctil | dsDNA, lineal | ϕMAM1 |
| Autographiviridae | cola descubierta, contráctil | dsDNA, lineal | Acintetobacter fago P2 | ||
| Myoviridae | cola descubierta, contráctil | dsDNA, lineal | T4 , Mu , P1 , Colifago P2 | ||
| Siphoviridae | cola descubierta, no contráctil (larga) | dsDNA, lineal | λ , T5 , HK97 , N15 | ||
| Podoviridae | cola descubierta, no contráctil (corta) | dsDNA, lineal | T7 , T3 , Φ29 , P22 | ||
| Monodnaviria | Haloruvirales | Pleolipoviridae | envuelto, pleomórfico | ssDNA , circular / dsDNA, circular / dsDNA lineal | HHPV1 , HRPV1 |
| Petitviral | Microviridae | descubierto, isométrico | ssDNA, circular | ΦX174 | |
| Tubulavirales | Inoviridae | descubierto, filamentoso | ssDNA, (en su mayoría) circular | M13 , CTXφ | |
| no asignado | Ligamento viral | Lipothrixviridae | envuelto, en forma de varilla | dsDNA, lineal | AFV1 |
| Rudiviridae | descubierto, en forma de varilla | dsDNA, lineal | SIRV1 | ||
| no asignado | no asignado | Ampullaviridae | envuelto, en forma de botella | dsDNA, lineal | ABV |
| Bicaudaviridae | descubierto, con forma de limón | dsDNA, circular | Canal de televisión británico | ||
| Clavaviridae | descubierto, en forma de varilla | dsDNA, circular | APBV1 | ||
| Finnlakeviridae | dsDNA | Voltear | |||
| Fuselloviridae | descubierto, con forma de limón | dsDNA, circular | SSV1 | ||
| Globuloviridae | envuelto, isométrico | dsDNA, lineal | PSV | ||
| Guttaviridae | descubierto, ovoide | dsDNA, circular | SNDV , APOV1 | ||
| Plasmaviridae | envuelto, pleomórfico | dsDNA, circular | Fago L2 | ||
| Portogloboviridae | envuelto, isométrico | dsDNA, circular | |||
| Spiraviridae | descubierto, en forma de varilla | ssDNA, circular | ACV | ||
| Tristromaviridae | envuelto, en forma de varilla | dsDNA, lineal | TTSV1 |
Los miembros de la familia Picobirnaviridae (orden Durnavirales ) también parecen infectar bacterias, no mamíferos.
Otra familia propuesta de fagos son los " Autolykiviridae " (dsDNA).
literatura
- Nicholas H. Mann: La tercera edad de los fagos. En: PLOS Biology . Volumen 3, No. 5, 17 de mayo de 2005, artículo e182, doi: 10.1371 / journal.pbio.0030182 ( texto completo en PDF ).
- Nancy Trun, Janine Trempy: bacteriófago. En: Nancy Jo Trun, JE Trempy, Janine Trempy: Genética bacteriana fundamental. Blackwell, Oxford 2003, ISBN 0-632-04448-9 ( texto completo en PDF ).
- Górski A, Weber-Dabrowska B: El papel potencial de los bacteriófagos endógenos en el control de patógenos invasores . En: Ciencias de la vida celular y molecular . Volumen 62, No. 5, marzo de 2005, págs. 511-519. doi : 10.1007 / s00018-004-4403-6 . PMID 15747058 .
- Forest Rohwer, Merry Youle, Heather Maughan, Nao Hisakawa, Leah L Pantéa: La vida en nuestro mundo de fagos: una guía de campo centenaria de los habitantes más diversos de la Tierra. Wholon, San Diega (CA) 2014, ISBN 978-0-9904943-0-0 .
- Hans Günther Schlegel, Georg Fuchs (Ed.): Microbiología general. 8a edición, Thieme, Stuttgart 2006, ISBN 3-13-444608-1 .
- Jong-Geol Kim, So-Jeong Kim, Virginija Cvirkaite-Krupovic, Mart Krupovic et al. : Los virus en forma de huso infectan la taumarchaea marina que oxida el amoniaco. En: Actas de la Academia Nacional de Ciencias . (PNAS) Volumen 116, No. 31, julio de 2019, artículo 201905682, doi: 10.1073 / pnas.1905682116 ( texto completo en línea ).
enlaces web
- Construye y multiplica con animación
- Bacteriófagos y terapia con fagos: preguntas y respuestas de un vistazo , información del Instituto Leibniz DSMZ (Colección Alemana de Microorganismos y Cultivos Celulares GmbH) en Braunschweig
- Instituto Federal de Evaluación de Riesgos (BfR) : preguntas y respuestas sobre bacteriófagos
- Sitio web del Instituto Eliava
- Terapia de fagos en el Instituto Ludwik Hirszfeld de Inmunología y Terapia Experimental, Breslau
- Terapia con fagos contra la neumonía , proyecto de investigación en París
- Phagoburn , proyecto de investigación de la UE sobre la terapia con fagos para las quemaduras
- "Comedores de bacterias" en lugar de antibióticos , VDI-Nachrichten, 25 de septiembre de 2015
- Healing Virus - Fighting Infections with Bacteriophages Radio broadcast, Bavaria 2, 29 de septiembre de 2012, consultado el 25 de septiembre de 2015
- Depredador bacteriano podría ayudar a reducir las muertes por COVID-19 - "Posible cambio de juego". El: '26 de junio de 2020, Fuente: Universidad de Birmingham
- Con virus fuera de la crisis de los antibióticos. - Documental de Jean Crépu sobre ARTE (F 2019, 54 min)
Evidencia individual
- ↑ NCBI: Bacillus phage Gamma (especie)
- ↑ a b Daniel Bojar: Útiles asesinos de bacterias. En: Spektrum.de - Spectrum of Science. desde junio de 2020, págs. 40–45.
- ↑ SIB: Virus que infectan a las bacterias. Activado: ViralZone .
- ↑ Mart Krupovic, Anja Spang, Simonetta Gribaldo, Patrick Forterre, Christa Schleper: Un provirus thaumarchaeal testifica una antigua asociación de virus de cola con arqueas. En: Transacciones de la Sociedad Bioquímica. Volumen 39, No. 1, enero de 2011, págs . 82-88, doi: 10.1042 / BST0390082 , PMID 21265751 .
- ↑ F. d'Hérelle (1917): Sur un microbe invisible antagonist des bacilles dysentériques. En: CR Ac. Ciencias. Núm. 165, págs. 373-375.
- ^ Loos-Frank, Brigitte, Lane, Richard P .: Biología de los parásitos . 3ª edición, actualizada y revisada. Springer Verlag, Berlín 2018, ISBN 978-3-662-54862-2 , págs. 4 ( google.de [consultado el 17 de marzo de 2019]).
- ↑ NCBI: Fagos T
- ↑ Rolf Sauermost, Doris Joyful et al. : Fago T. - Léxico de biología. En: Spektrum.de , consultado el 31 de enero de 2021. Las asignaciones familiares ya no corresponden al estado actual según ICTV.
- ↑ L. McKane, J. J. Ferretti: interacciones fago-huésped y la producción de tipo A de exotoxina estreptococos del grupo A, estreptococos . En: Infección e inmunidad . Volumen 34, No. 3, diciembre de 1981, págs. 915-919. PMID 7037644 . PMC 350956 (texto completo gratuito).
- ↑ Victor Krylov, Maria Bourkaltseva, Elena Pleteneva, Olga Shaburova, Sergey Krylov, Alexander Karaulov, Sergey Zhavoronok, Oxana Svitich, Vitaly Zverev; Julie Thomas, Lindsay Black (Eds.): Phage phiKZ - El primero de los gigantes. En: Virus. Volumen 13, N ° 2, Edición especial Fagos gigantes o gigantes. 149, 20 de enero de 2021, doi: 10.3390 / v13020149 .
- ↑ a b Audra E. Devoto, Joanne M. Santini y col. : Los megafagos infectan a Prevotella y las variantes están muy extendidas en los microbiomas intestinales. En: Nature Microbiology. Volumen 4, págs.693-700, 28 de enero de 2019, doi: 10.1038 / s41564-018-0338-9 , especialmente la Tabla 1 y la Figura complementaria 11 .
- ↑ Lakshminarayan M. Iyer, Vivek Anantharaman, Arunkumar Krishnan, A. Maxwell Burroughs, L. Aravind: Jumbo Phages: A Comparative Genomic Overview of Core Functions and Adaptions for Biological Conflicts , en: MDPI Virus, Volumen 13, No. 1, Especial Issue Giant or Jumbo Phages, 5 de enero de 2021, pág. 63; doi: 10.3390 / v13010063 , especialmente suplemento (zip con PDF y xlsx)
- ↑ NCBI: fago de Sphingomonas PAU. (especies)
- ↑ Richard Allen White III, Curtis A. Suttle: El borrador de la secuencia del genoma de la cepa HER1398 (Proteobacteria) de 'Sphingomonas paucimobilis', hospedante del fago PAU gigante, indica que es un miembro del género 'Sphingobacterium' (Bacteroidetes). En: Anuncios del genoma. Volumen 1, No. 4, julio-agosto de 2013, e00598-13, doi: 10.1128 / genomeA.00598-13 , PMID 23929486 , PMC 3738902 (texto completo libre)
- ^ Universidad de California - Berkeley: [1] , ScienceDaily, 28 de enero de 2019.
- ↑ UCL: Nuevo virus bacteriano gigante encontrado en el intestino humano. University College London, 29 de enero de 2019.
- ^ Colm Gorey: Los virus gigantes descubiertos en humanos plantean preguntas sobre la vida misma. En: siliconrepublic.com a partir del 29 de enero de 2019.
- ↑ a b Basem Al-Shayeb, Rohan Sachdeva, L. Chen, Jillian F. Banfield et al. : Clados de fagos enormes de todos los ecosistemas de la Tierra , en: Nature del 12 de febrero de 2020, doi: 10.1038 / s41586-020-2007-4 , bioRxiv : 10.1101 / 572362v1 ( preimpresión del texto completo)
- ↑ Ed Yong: Un gran descubrimiento en el mundo de los virus. En: El Atlántico. a partir del 20 de febrero de 2020.
- ↑ Michael Le Page: Los virus gigantes han convertido a CRISPR en armas contra sus huéspedes bacterianos. Activado: NewScientist. a partir del 30 de marzo de 2019.
- ↑ Los bacteriófagos gigantes abren la brecha entre los microbios vivos y las máquinas virales. On: SCI-NEWS a partir del 13 de febrero de 2020.
- ↑ Tessa Koumoundouros: Los científicos descubren virus gigantes con características que solo se han visto antes en células vivas. On: ScienceAlert del 14 de febrero de 2020.
- ↑ Daniela Albat: Fago con genoma de tamaño récord descubierto , en: scinexx.de del 18 de febrero de 2020
- ↑ Jan Osterkamp: Se supone que Anti-CRISPR mejora CRISPR. En: Spektrum.de a partir del 16 de enero de 2020.
- ↑ Annika Röcker: Las tijeras genéticas son impotentes contra algunos virus. En: Spektrum.de a partir del 10 de diciembre de 2019.
-
↑ Basem Al-Shayeb, Rohan Sachdeva, Lin-Xing Chen, Cindy J. Castelle, Alexander L. Jaffe, Jennifer A. Doudna , Jillian F. Banfield et al. : Clados de fagos enormes de todos los ecosistemas de la Tierra. En: Naturaleza . No. 578, 12 de febrero de 2020, págs. 425-431, doi: 10.1038 / s41586-020-2007-4 . Además:
- bioRxiv , PrePrint del 11 de marzo de 2019, doi: 10.1101 / 572362
- Los enormes virus que se alimentan de bacterias cierran la brecha entre la vida y la no vida: los grandes bacteriófagos portan genes bacterianos, incluidas las proteínas CRISPR y ribosomales. En: eurekalert.org a partir del 12 de febrero de 2020.
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas et al.: Virus encontrado en un lago boreal vincula virus ssDNA y dsDNA. En: Actas de la Academia Nacional de Ciencias. Volumen 114, No. 31, julio de 2017, doi: 10.1073 / pnas.1703834114 .
- ↑ a b Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang et al.: Un linaje principal de virus dsDNA sin cola como asesinos no reconocidos de bacterias marinas. En: Naturaleza. Volumen 554, págs.118-122, 24 de enero de 2018, doi: 10.1038 / nature25474 .
- ^ Los científicos encuentran un nuevo tipo de virus en los océanos del mundo: Autolykiviridae. On: sci-news del 25 de enero de 2018.
- ↑ David L. Chandler: Investigadores descubren un eslabón perdido en la evolución del virus. En: SciTechDaily del 25 de enero de 2018.
- ↑ Los investigadores descubren un virus misterioso que domina los océanos. Activado: información privilegiada de la empresa desde el 29 de enero de 2018.
- ↑ Se acaban de descubrir en el océano virus nunca antes vistos con ADN extraño. En: sciencealert.com a partir del 25 de enero de 2018.
- ↑ NCBI: Autolykiviridae. (familia) - virus dsDNA no clasificados.
- ↑ E-Bin Gao, Xiu-Ping Yuan, Ren-hui Li, Qi-Ya Zhang: Aislamiento de un nuevo cianófago infeccioso para la cianobacteria filamentosa "Planktothrix agardhii" (Cyanophyceae) del lago Donghu, China. En: Ecología microbiana acuática. (AME), Volumen 54, No. 1, febrero de 2009, págs. 163-170, doi: 10.3354 / ame01266 ( texto completo en PDF ).
- ↑ Mélanie Gerphagnon, Deborah J. Macarthur, Delphine Latour, Claire M. M. Gachon, Floris Van Ogtrop, Frank H. Gleason, Télesphore Sime - Ngando: Agentes microbianos involucrados en el declive de las floraciones de cianobacterias filamentosas y coloniales con un enfoque en el parasitismo fúngico. En: sfam Environmental Microbiology. Volumen 17, No. 8, págs. 2573-2587, doi: 10.1111 / 1462-2920.12860 ( texto completo en PDF ), en particular un suplemento; Texto completo en PDF Tabla 2.
- ^ NCBI: fago de Planktothrix PAV-LD. (especies)
- ↑ CrAssphage: El virus del intestino antiguo previamente desconocido vive en la mitad de la población mundial. On: sci-news del 11 de agosto de 2014 ( inglés ).
- ↑ Natalia Yutin, Kira S. Makarova, Ayal B. Gussow, Mart Krupovic, Anca Segall, Robert A. Edwards, Eugene V. Koonin: Descubrimiento de una familia de bacteriófagos expansiva que incluye los virus más abundantes del intestino humano . En: Nature Microbiology . 3, núm. 1, 2017, págs. 38–46. doi : 10.1038 / s41564-017-0053-y . PMID 29133882 . PMC 5736458 (texto completo gratuito).
- ↑ Eugene V. Koonin: Detrás del artículo: El virus asociado con humanos más abundante ya no es huérfano , 13 de noviembre de 2017
- ↑ SIB: fagos crAsslike. En: viralzone.expasy.org - Orden: Caudovirales , estimado en 10 géneros.
-
↑ Luis Fernando Camarillo-Guerrero, Alexandre Almeida, Guillermo Rangel-Pineros, Robert D. Finn, Trevor D. Lawley: Expansión masiva de la diversidad de bacteriófagos intestinales humanos , en: Cell Resource Volume 184, No. 4, pp. 1098-1109. e9, 18 de febrero de 2021, doi: 10.1016 / j.cell.2021.01.029 . PrePrint del 3 de septiembre de 2020: bioRxiv , Europa PMC , doi: 10.1101 / 2020.09.03.280214 . Además:
- Martin Vieweg: Se han descubierto miles de tipos de virus en la flora intestinal , On: Wissenschaft.de del 18 de febrero de 2021 (alemán)
- Daniel Lingenhöhl: Virobiom : Gut es el hogar de decenas de miles de tipos desconocidos de virus , en: Spektrum.de desde el 19 de febrero de 2021 (alemán)
- Los biólogos encuentran casi 143.000 especies de bacteriófagos en el intestino humano. On: sci-news del 19 de febrero de 2021
- Peter Dockrill: Los científicos encuentran 140.000 especies de virus en el intestino humano y la mayoría son desconocidas. En: sciencealert.com a partir del 28 de febrero de 2021
- Los científicos identifican más de 140.000 especies de virus en el intestino humano. En: sciencedaily.com a partir del 18 de febrero de 2021
- ↑ Luis Fernando Camarillo Guerrero: Análisis integrador del fageoma del intestino humano utilizando un enfoque metagenómico. Tesis de doctorado, Gonville & Caius College, Universidad de Cambridge, agosto de 2020, doi: 10.17863 / CAM.63973 .
- ^ Luego Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: una hoja de ruta para la taxonomía de fagos basada en el genoma. En: Virus MDPI. Volumen 13, No. 3, Sección Virus bacterianos, 18 de marzo de 2021, p. 506, doi: 10.3390 / v13030506 .
- ↑ Nina S. Atanasova, Hanna M. Oksanen, Dennis H. Bamford : Halovirus de arqueas, bacterias y eucariotas. En: Opinión actual en microbiología. 25 de junio de 2015, págs. 40-48, doi: 10.1016 / j.mib.2015.04.001 , PMID 25932531 .
- ↑ NCBI: Halovirus (clado)
- ↑ NCBI: Myohalovirus (género)
- ^ ICTV: Historia de la taxonomía de ICTV: virus de Halobacterium phiH
- ↑ NCBI: Fago Halorubrum HF2 (especie)
- ↑ a b c Yosuke Nishimura, Hiroyasu Watai, Takashi Honda et al.: Los genomas virales ambientales arrojan nueva luz sobre las interacciones virus-huésped en el océano. En: mSphere. Volumen 2, No. 2, marzo - abril de 2017, e00359-16, doi: 10.1128 / mSphere.00359-16 , PMC 5332604 (texto completo libre), PMID 28261669 , especialmente Figura 4.
- ↑ a b c Darius Kazlauskas, Mart Krupovic, Česlovas Venclovas: La lógica de la replicación del ADN en virus de ADN bicatenario: conocimientos del análisis global de genomas virales. En: Investigación de ácidos nucleicos. Volumen 44, No. 10, 2 de junio de 2016, págs. 4551–4564, doi: 10.1093 / nar / gkw322 , PMC 4889955 (texto completo libre), PMID 27112572 .
- ↑ a b c D Prangishvili, DH Bamford, P Forterre, J Iranzo, EV Koonin, M Krupovic: The enigmatic archaeal virosphere . En: Nature Reviews Microbiology . Volumen 15, No. 12, 10 de noviembre de 2017, págs.724-739. doi : 10.1038 / nrmicro.2017.125 . PMID 29123227 . Ver en particular la FIG.1
- ↑ NCBI: Halovirus HSTV-1 (especie)
- ↑ NCBI: Halovirus HSTV-2 (especie)
- ↑ NCBI: Halovirus HVTV-1 (especie)
- ↑ Anukriti Sharma, Matthias Schmidt, Bärbel Kiesel et al.: Virus bacterianos y arqueales de las aguas termales del Himalaya en Manikaran Modulate Host Genomes. En: Fronteras en Microbiología. Volumen 9, 14 de diciembre de 2018, p. 3095, doi: 10.3389 / fmicb.2018.03095 , PMC 6302217 (texto completo gratuito), PMID 30619174 ( texto completo como PDF ).
- ↑ SIB: Fuselloviridae. En: viralzone.expasy.org .
- ↑ NCBI: Halovirus VNH-1 (especie)
- ↑ Carolina M. Mizuno, Bina Prajapati, Soizick Lucas - Staat, Telesphore Sime - Ngando, Patrick Forterre, Dennis H. Bamford, David Prangishvili, Mart Krupovic, Hanna M. Oksanen: Nuevos virus haloarqueales del lago Retba que infectan las especies Haloferax y Halorubrum. En: Microbiología ambiental. Volumen 21, No. 6, sfam, 28 de marzo de 2019, doi: 10.1111 / 1462-2920.14604 .
- ↑ NCBI: Halo virus , Halo Virus
- ↑ a b c Alon Philosof, Natalya Yutin, José Flores-Uribe, Itai Sharon, Eugene V. Koonin, Oded Béjà: Nuevos virus oceánicos abundantes del grupo marino no cultivado II Euryarchaeota. En: Biología actual. Volumen 27, No. 9), 8 de mayo de 2017, págs. 1362-1368, doi: 10.1016 / j.cub.2017.03.052 , PMC 5434244 (texto completo libre), PMID 28457865 .
- ↑ Luis H. Orellana, T. Ben Francis, Karen Krüger, Hanno Teeling, Marie-Caroline Müller, Bernhard M. Fuchs, Konstantinos T. Konstantinidis, Rudolf I. Amann: diferenciación de nicho entre el grupo marino costero II Euryarchaeota recurrente anualmente. En: Nature ISME Journal. Volumen 13, 26 de agosto de 2019, págs.3014-3036, doi: 10.1038 / s41396-019-0491-z .
- ↑ Xiaomin Xia, Wang Guo, Hongbin Liu: Variación de la escala de la cuenca en la composición y diversidad de las arqueas en el Océano Pacífico. En: Fronteras en Microbiología. 23 de octubre de 2017, doi: 10.3389 / fmicb.2017.02057 .
- ↑ Ana-Belen Martin-Cuadrado et al. : Una nueva clase de Euryarchaeota marina del grupo II del máximo mediterráneo de clorofila profunda. En: Nature ISME Journal. Volumen 9, 2015, págs. 1619-1634, doi: 10.1038 / ismej.2014.249 .
- ↑ Daria Vaisman: Cómeme . Disponible en : slate.com desde mayo de 2006.
- ↑ Bettina Hofer: Conservación con virus. Heise Technology Review, 28 de febrero de 2013, consultado el 7 de agosto de 2014 .
- ↑ JW Kretzer, R. Lehmann et al.: Uso de dominios de unión a la pared celular de alta afinidad de endolisinas de bacteriófagos para la inmovilización y separación de células bacterianas. En: Microbiología aplicada y ambiental. Volumen 73, 2007, págs. 1992-2000.
- ↑ C. Rozand, PCH Feng: Análisis de especificidad de un nuevo ligando derivado de fagos en un ensayo fluorescente ligado a enzimas para la detección de Escherichia coli O157: H7. J. En: protección alimentaria. Volumen 72, 2009, págs. 1078-1081.
- ↑ Bacteriophages - New Applications in Food Microbiology ( Memento del 2 de marzo de 2013 en Internet Archive ) bioFood n ° 3 de diciembre de 2006, p. 2.
- ↑ Aplicaciones de la tecnología de ligandos de fagos (eliminación de endotoxinas, detección de endotoxinas, pruebas de calidad alimentaria). En: hyglos.de .
- ↑ DM Guglielmotti, DJ Mercanti, JA Reinheimer, ADL Quiberoni: Eficiencia de tratamientos físicos y químicos sobre la inactivación de bacteriófagos lácteos. En: Fronteras en Microbiología. Volumen 2, 2012, doi: 10.3389 / fmicb.2011.00282 .
- ↑ Renata Filipa Cruz de Matos: Profagos de Enterococcus faecalis V583: Interacciones dinámicas y contribución a los rasgos patógenos bacterianos , disertación, Universidade Nova de Lisboa (UNL), julio de 2013
- ↑ NCBI: satélite Phage P4 (sin rango)
- ↑ SIB: 20 (Fuselloviridae) . En: viralzone.expasy.org .
- ↑ SIB: Salterprovirus. 190 Provirus de Salter. , En: viralzone.expasy.org .
- ↑ NCBI: Virus His 1 (especie)
- ↑ SIB: Bicaudaviridae. En: viralzone.expasy.org .
- ↑ ICTV: Historia de la taxonomía de ICTV: Nitmarvirus NSV1 , EC 51, Berlín, Alemania, julio de 2019; Ratificación por correo electrónico de marzo de 2020 (MSL # 35)
- ↑ SIB: 113 (Inoviridae) en: viralzone.expasy.org .
- ↑ Stephen Nayfach, Simon Roux, Emiley A. Eloe-Fadrosh et al. : Un catálogo genómico de los microbiomas de la Tierra. En: Naturaleza . Biotecnología, 6 de noviembre de 2020, doi: 10.1038 / s41587-020-0718-6 , aquí: Figura S13 B.
- ↑ ICTV: Historia de la taxonomía de ICTV: Gammapleolipovirus His2 , EC 51, Berlín, Alemania, julio de 2019; Ratificación por correo electrónico de marzo de 2020 (MSL # 35)
- ↑ S. McGrath, D. van Sinderen D (Ed.): Bacteriófago: Genética y Biología Molecular , 1er. Edición, Caister Academic Press, 2007, ISBN 978-1-904455-14-1 .
- ↑ isométrico para partículas de virus : aproximadamente la misma expansión espacial en cada dirección, por ejemplo , esférica o icosaédrica .
- ↑ SIB: Ampullaviridae. En: viralzone.expasy.org .
- ↑ SIB: Bicaudaviridae. En: viralzone.expasy.org .
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum, David I. Stuart, Janne J. Ravantti, Juha Huiskonen, Lotta-Riina Sundberg: Virus encontrado en un lago boreal vincula virus ssDNA y dsDNA . En: Actas de la Academia Nacional de Ciencias. Volumen 114, No. 31, julio de 2017, doi: 10.1073 / pnas.1703834114 .
- ↑ SIB: Fuselloviridae. En: viralzone.expasy.org .
- ↑ SIB: Globuloviridae. En: viralzone.expasy.org .
- ↑ S. R. Krishnamurthy, D. Wang: Amplia conservación de los sitios de unión ribosomal procarióticos en picobirnavirus conocidos y nuevos . En: Virología . Volumen 516, 2018, págs. 108-114. doi : 10.1016 / j.virol.2018.01.006 . PMID 29346073 .