La transcriptasa inversa
| La transcriptasa inversa | ||
|---|---|---|
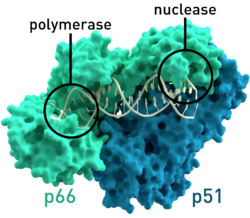
| ||
| Modelo de transcriptasa inversa (dímero) del VIH- 1 | ||
| Clasificación de enzimas | ||
| CE, categoría | 2.7.7.49 , nucleotidil transferasa | |
| Sustrato | Trifosfato de desoxinucleósido + ADN (s) | |
| Productos | Difosfato + ADN (n + 1) | |
Las transcriptasas inversas ( RT ) son proteínas enzimáticamente activas conocidas como ADN polimerasas dependientes de ARN una transcripción , es decir, en la dirección inversa (inversa) del ARN en el ADN catalizado ; esto permite que la información genética se transcriba del ARN al ADN.
Aspectos bioquímicos
Por medio de su actividad de ADN polimerasa dependiente de ARN, primero se construye una doble hebra híbrida de ARN y ADN después de que se haya presentado un ARN monocatenario mediante la unión de bloques de construcción de ADN complementarios emparejados ( desoxirribonucleótidos ). A partir de entonces, su porción de ARN se descompone en gran medida por medio de una actividad de ARNasa H de una sección particular de la proteína. La hebra única restante de ADN finalmente se complementa para formar una doble hebra de ADN, catalizada por una actividad adicional de la ADN polimerasa dependiente del ADN inherente de la transcriptasa inversa.
La tasa de error de la transcriptasa inversa debido a la falta de corrección de pruebas es de 1:10 3 a 1:10 4 y conduce a una tasa de mutación muy alta .
historia
La transcriptasa inversa de los retrovirus fue descrita por primera vez en 1970 tanto por Howard Temin como independiente de David Baltimore . En 1975, usted y Renato Dulbecco recibieron el Premio Nobel de Fisiología o Medicina por este descubrimiento . La adición inversa denota la capacidad peculiar de esta enzima para construir un ADN basado en una plantilla de ARN. La dirección inesperada del proceso invalida la doctrina previamente sostenida, el llamado dogma central de la biología molecular , de que el flujo de información genética corre exclusivamente en la dirección de ADN → ARN → proteína, nunca al revés.
Ocurrencia
La transcriptasa inversa se descubrió por primera vez en retrovirus (por ejemplo , VIH , HTLV , SIV ). Estos virus con un genoma de ARN usan RT para reescribir su genoma en el ADN. El RT cumple así una función crucial en la multiplicación del virus. Además, ciertos virus de ADN como los hepadnavirus (por ejemplo, el patógeno que causa la hepatitis B ( VHB, la proteína P ) o los caulimovirus que se encuentran en las plantas ) también contienen un RT. Los transposones de clase I , también llamados retroelementos , también se derivan de antiguos retrovirus mutados . Estos necesitan un RT para su replicación. Esto lo codifica usted ( LINE autónomos y retrotransposones LTR ) o debe estar disponible (por ejemplo, con SINE ).
Los intrones del grupo II también codifican una transcriptasa inversa, que estabiliza un ARN intrón enzimáticamente activo ( ribozima ) y transcribe el ARN integrado en ADN. El ARN cataliza el empalme en el proceso. Se han detectado intrones del grupo II en procariotas y en los genomas de orgánulos en hongos y plantas.
Una transcriptasa inversa también forma parte de la telomerasa de los eucariotas , donde expande los telómeros, que se han acortado en el curso de la replicación , a su longitud original y, por lo tanto, retrasa el proceso de envejecimiento celular . El nombre más preciso es telomerasa transcriptasa inversa (TERT), como. B. para la transcriptasa inversa de la telomerasa humana (hTERT).
Transcripción inversa
El genoma de ARN de los virus de ARN, por ejemplo los retrovirus , se transcribe en ADN de doble hebra. Este proceso se llama transcripción inversa . El virus trae consigo la transcriptasa inversa necesaria en sus partículas de virus. Con la ayuda de un cebador de ARNt , este primero transcribe el ARN monocatenario del virus en una cadena de ADN complementaria (actividad como ADN polimerasa dependiente de ARN ). Luego, el ARN se descompone en un fragmento que sirve como segundo cebador (actividad como ribonucleasa H ). Esto luego crea el ADN de doble hebra (actividad como ADN polimerasa dependiente de ADN ).
El genoma viral del ARN está disponible como una copia de ADN de doble hebra. Además, se generaron las denominadas secuencias LTR en ambos extremos de las cadenas de ADN durante la transcripción inversa , que son esenciales para el curso posterior de la infección. Permiten la integración en el genoma del ADN de la célula huésped, utilizando otra enzima de retrovirus, una integrasa .
Aplicaciones biotecnológicas
La frecuencia de errores en la transcriptasa inversa viral dificulta la lucha contra retrovirus como el VIH. La inhibición de la transcriptasa inversa es un objetivo de la terapia de combinación y es posible a través de varios principios activos ( NNRTI , NRTI ). Estos inhibidores de la transcriptasa inversa fueron los primeros y hasta 1994 los únicos fármacos eficaces aprobados para el tratamiento de la infección por VIH.
La transcriptasa inversa generada artificialmente "Xenopolimerasa" ahora puede corregir y minimizar así la tasa de error.
Las transcriptasas inversas se utilizan en biología molecular y diagnóstico molecular , por ejemplo en RT-PCR o para crear un banco de ADNc . Para ello se utilizan transcriptasas inversas virales del virus de la leucemia murina (MLV) o del virus de la mieloblastosis aviar (AMV). Sin embargo, por regla general, no se utilizan enzimas nativas, sino variantes diseñadas genéticamente con una tasa de error más baja, una actividad de RNasa H más baja y una estabilidad de temperatura más alta. Después de que el aislamiento de las transcriptasas inversas del intrón recombinante del grupo II haya tenido éxito, se espera que, debido a su menor tasa de error, mayor velocidad y estabilidad de temperatura, pero en particular también la actividad de cambio de plantilla, el cambio directo al extremo 3 'de nuevos ARN, ganan en importancia.
Evidencia individual
- ^ Racaniello, VR (Vincent R.), Rall, Glenn F., Skalka, Anna Marie, Enquist, LW (Lynn W.),: Principios de virología . 4ª edición. Washington, DC, ISBN 978-1-55581-933-0 , págs. 189 .
- ↑ a b Creación de transcriptasa inversa con capacidad de corrección. Consultado el 27 de febrero de 2020 .
- ↑ HM Temin, S. Mizutani: ADN polimerasa dependiente de ARN en viriones del virus del sarcoma de Rous. En: Naturaleza. Volumen 226, Número 5252, junio de 1970, págs. 1211-1213, PMID 4316301 .
- ↑ D. Baltimore: ADN polimerasa dependiente de ARN en viriones de virus tumorales de ARN. En: Naturaleza. Volumen 226, Número 5252, junio de 1970, págs. 1209-1211, PMID 4316300 .
- ^ John M. Coffin, Stephen H. Hughes, Harold E. Varmus : El lugar de los retrovirus en biología. En: Retrovirus. Prensa de laboratorio de Cold Spring Harbor, 1997, ISBN 0-87969-571-4 .
- ↑ Dogma central invertido. En: Naturaleza. Volumen 226, Número 5252, junio de 1970, págs. 1198-1199, PMID 5422595 .
- ↑ AM Lambowitz, S. Zimmerly: Intrones del grupo II: ribozimas móviles que invaden el ADN. En: Perspectivas de Cold Spring Harbor en biología. Volumen 3, número 8, agosto de 2011, p. A003616, doi: 10.1101 / cshperspect.a003616 , PMID 20463000 , PMC 3140690 (texto completo gratuito) (revisión).
- ↑ Jeremy Cherfas: Hayflick Licked: Telomerase alarga la vida de las células humanas normales ( Memento del 8 de agosto de 2012 en Internet Archive )
- ^ Witzany G (agosto de 2008). Los orígenes virales de los telómeros y las telomerasas y su importante papel en la eucariogénesis y el mantenimiento del genoma. (PDF; 263 kB). En: Biosemiotics 1 (2): 191-206. doi: 10.1007 / s12304-008-9018-0
- ↑ MJ Wacker, MP Godard: Análisis de RT-PCR en tiempo real de uno y dos pasos usando SuperScript III. En: Revista de técnicas biomoleculares: JBT. Volumen 16, Número 3, septiembre de 2005, págs. 266-271, PMID 16461951 , PMC 2291734 (texto completo libre).
- ↑ A. Baranauskas, S. Paliksa, G. Alzbutas, M. Vaitkevicius, J. Lubiene, V. Letukiene, p Burinskas, G. Snauvicius, R. Skirgaila: Generación y caracterización de nueva mesa procesiva altamente termoestable y reverso M-MuLV variantes de transcriptasa. En: Ingeniería, diseño y selección de proteínas: PEDS. Volumen 25, Número 10, octubre de 2012, págs. 657-668, doi: 10.1093 / protein / gzs034 , PMID 22691702 .
- ^ S. Mohr, E. Ghanem, W. Smith, D. Sheeter, Y. Qin, O. King, D. Polioudakis, VR Iyer, S. Hunicke-Smith, S. Swamy, S. Kuersten, AM Lambowitz: termoestable proteínas de fusión de la transcriptasa inversa del intrón del grupo II y su uso en la síntesis de ADNc y la secuenciación de ARN de próxima generación. En: ARN. Volumen 19, número 7, julio de 2013, págs. 958-970, doi: 10.1261 / rna.039743.113 , PMID 23697550 , PMC 3683930 (texto completo libre).
enlaces web
- La transcriptasa inversa del VIH
- Presentación interactiva de la RT del VIH en el Museo Macromolecular en línea (el archivo PDB se muestra a través de un visor de JavaScript)
